- トップページ
- NEWS & TOPICS
- 岡田 啓汰さんらの論文が英国科学雑誌「Nucleic Acids Researc…
岡田 啓汰さん(大学院修士課程生命科学コース 2021年修了)、青木 伽奈枝さん(同コース 2年・広島市立美鈴が丘高等学校出身/指導教員:分子発生制御学研究室 蒲池 雄介教授)らの論文が英国科学雑誌「Nucleic Acids Research」に掲載されました。
「Nucleic Acids Research」は1974年にオックスフォード大学出版局によって発行されたオープンアクセスの査読つき科学ジャーナルであり、DNAやRNAなどの核酸に関する研究とそれらに関連する研究論文が掲載されています。
岡田さんらが発表した論文のタイトルは「Key sequence features of CRISPR RNA for dual-guide CRISPR-Cas9 ribonucleoprotein complexes assembled with wild-type or HiFi Cas9(dual-guide CRISPR RNAと野生型またはHiFi Cas9からなるリボヌクレオタンパク質複合体の切断効率に影響を与えるcrRNAの配列特徴)」です。
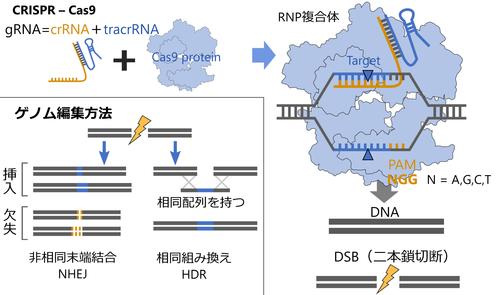
ゲノム編集は、DNA二本鎖切断後の生体内修復機能を利用して行われています。この二本鎖切断を誘導するツールとして、現在 CRISPR-Cas9(clustered regularly interspaced short palindromic repeats / CRISPR associated proteins)※が主に使用されています。 DNA二本鎖切断活性を持つCRISPR-Cas9 リボヌクレオタンパク質 (RNP)複合体は、CRISPR RNA(crRNA)と trans-activating crRNA(tracrRNA)の二つの短いRNAからなる guide RNA(gRNA)とCas9タンパク質から形成されています。CRISPR-Cas9 RNPによる標的DNAの認識は、Cas9タンパク質がNGGというPAM配列と相互作用し、次にcrRNAがPAM配列に隣接する相補的なDNAに結合することで行われています。 crRNAの配列を変更するだけで、ゲノム上の狙った場所を切断できるという使いやすさから CRISPR Cas9はゲノム編集において重要なツールとなっています。
しかし、crRNA配列により切断効率が異なることが知られており、ゲノム編集の成功には切断効率の高いcrRNAの選択が重要となります。従来はcrRNAと tracrRNAを融合したsingle guide RNA(sgRNA)が広く利用されていましたが、in vitroあるいはin vivo転写反応により合成される sgRNAは 5'末端がグアニン(G)になるため標的配列の選択に制限をかけるだけではなく、CRISPR-Cas9の切断効率を大幅に低下させることが問題となっています。これまでの研究では、ほとんどの場合sgRNAが用いられていたため、crRNAの配列特徴と切断効率の関係が不明確なままでした。
(図1 一般的なゲノム編集方法とCRISPR-Cas9の切断方法)
そこで、岡田さんらは、sgRNAに関わる制限がない、二つのRNAを合わせて用いるdual-guide RNA(dgRNA)を用いて、crRNAの塩基配列と切断効率の関係を調べることにしました。
この実験では、ゼブラフィッシュ(Danio rerio)ゲノム上の 17 個の遺伝子に対して 51 個の crRNA を設計し、切断効率を測定しました。まず、野生型のSpCas9(WT Cas9)タンパク質あるいは特異性が向上した SpCas9 バリアントである High Fidelity SpCas9(HiFi Cas9)タンパク質から構成される RNP 複合体を受精直後のゼブラフィッシュ胚に顕微注入し、次の日に胚のゲノム DNA を調製しました。CRISPR-Cas9による切断が起こると、DNA修復の過程で配列の欠失や挿入(インデル)が生じます。インデルが生じている割合を調べることで、それぞれのcrRNAの切断効率の違いがわかります。これを調べるために、標的配列をPCR法で増幅し、その塩基配列をサンガーシーケンスで分析します。PCR産物は、もとの配列に加えて様々なインデルを含んでいます。したがって、そこから得られた波形データをもとの配列の波形データと比較することで、どの程度インデルが生じているかがわかります。
この分析には、Tracking of Indels by Decomposition(TIDE)および Inference of CRISPR Editing(ICE)と呼ばれるコンピューターのプログラムを用いました。このようにして得られた51 個のcrRNA の切断効率と標的配列を k-mer probability logo(kpLogo)と呼ばれる別のコンピューターのプログラムを用いて分析し、切断効率に応じた各位置における塩基の出現頻度の統計的有意性をP値として算出しました。この解析をおこなうことで、crRNAの各塩基が切断の効率を上げるのか、あるいは下げるのかの特徴が明らかになりました(図2には、P値を-log10(P)に変換した値に応じた大きさの塩基ロゴとして表しています)。
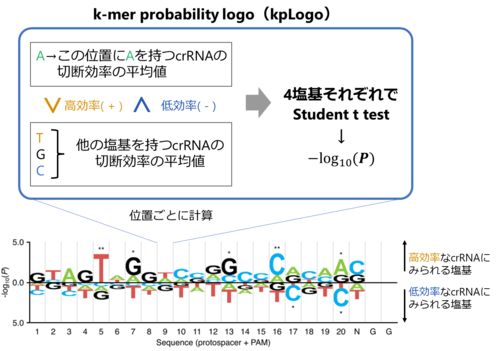
(図2 kp-Logoの計算方法と得られた位置特異的配列特徴)
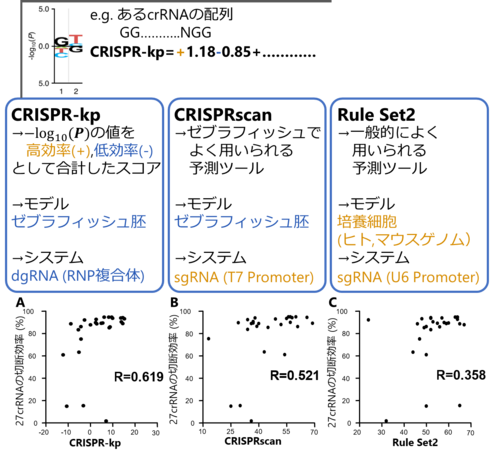
岡田さんの研究を引き継いだ青木さんは、51個のcrRNAの切断効率より得られたポジションごとの各塩基のP値が、別のcrRNAの活性予測に使えるかどうかを、新たに設計した27個のcrRNAを用いて調べました 。活性予測には、切断の効率を上げる塩基の-log10(P)値をプラス値として、効率を下げる塩基の場合はマイナス値として全ポジションの総和を求め、それをCRISPR-kpスコアとして用いました。
その結果、実測した27個のcrRNAの切断効率はCRISPR-kp予測値との相関が見られました。既存の予測ツールとの比較では、一般的によく用いられている予測ツールである Doench Rule Set 2や、ゼブラフィッシュ胚でsgRNAを用いたデータに基づくCRISPRscanのスコアよりも予測精度が高くなりました。
(図3 新しく設計した27crRNAの切断効率と予測スコアとの比較)
本研究により、crRNAの塩基配列と切断効率の関係が格段に明瞭になりました。この成果は、ゼブラフィッシュばかりでなく、他の生物種、さらに医学的応用に向けたゲノム編集技術の高度化に貢献することが期待されます。
※1 CRISPR-Cas9(clustered regularly interspaced short palindromic repeats / CRISPR associated proteins)
DNA二本鎖を切断(Double Strand Breaks=DSBs)してゲノム配列の任意の場所を削除、置換、挿入することができる新しい遺伝子改変技術"ゲノム編集"で用いられる主要なツールです。ZFN、TALENに続く第3世代のゲノム編集ツールとして2013年に報告されたCRISPR-Cas技術は、カスタム化(標的遺伝子の変更や複数遺伝子のターゲット)が容易であることから、現在、ヒトやマウスといった哺乳類細胞ばかりではなく、細菌、寄生生物、ゼブラフィッシュなどの膨大な種類の細胞や生物種において、そのゲノム編集に急速に利用が広まっています。
【掲載論文】
題 名:Key sequence features of CRISPR RNA for dual-guide CRISPR-Cas9 ribonucleoprotein complexes assembled with wild-type or HiFi Cas9 (dual-guide CRISPR RNAと野生型またはHiFi Cas9からなるリボヌクレオタンパク質複合体の切断効率に影響を与えるcrRNAの配列特徴)
掲載誌:Nucleic Acids Research
掲載日:2022年2月15日
著者名:
高知工科大学大学院修士課程 生命科学コース 2021年修了 岡田 啓汰
高知工科大学大学院修士課程 生命科学コース 2年 青木 伽奈枝
高知工科大学大学院修士課程 生命科学コース 2022年修了 田部井 輝侑
高知工科大学大学院修士課程 生命科学コース 2年 椙尾 浩大
高知工科大学大学院修士課程 生命科学コース 2021年修了 今井 捷智
高知工科大学大学院修士課程 生命科学コース 2022年修了 盆子原 友希
高知工科大学 環境理工学群 教授 蒲池 雄介
DOI:https://doi.org/10.1093/nar/gkac100
RELATED POST
関連記事